Le réseau de neurones accélère la reconstruction de l’image 3D d’échantillons biologiques
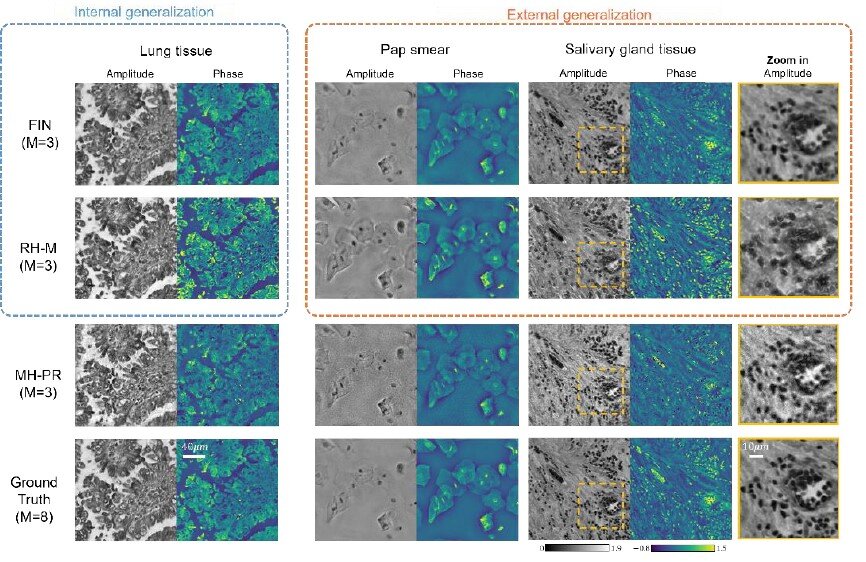
Comparaison de la circularisation interne et externe de FIN et RH-M sur des coupes de tissu pulmonaire, des glandes salivaires et des échantillons de frottis Pap. Les résultats de la reconstruction MH-PR utilisant les mêmes hologrammes d’entrée (M = 3) sont également présentés à titre de comparaison. Crédit : Hanlong Chen, UCLA
Les chercheurs ont développé un nouveau réseau neuronal complet qui peut accélérer la reconstruction des images 3D. Contrairement à d’autres techniques d’apprentissage en profondeur, cette approche peut être utilisée sur des échantillons qui n’ont pas été rencontrés pendant la formation, ce qui la rend particulièrement utile pour l’imagerie biomédicale 3D sans étiquette.
« Avec ce cadre, bien formé réseau neuronal Ils peuvent être distribués n’importe où, sans réglage fin, et effectuer une imagerie 3D rapide et de haute qualité de différents échantillons », a expliqué le directeur de recherche Hanlong Chen, Université de Californie, Los Angeles (UCLA).
Hanlong Chen et Aydogan Ozkan présenteront la recherche à la conférence Frontiers in Optics + Laser Sciences (FiO LSLa réunion a eu lieu à Rochester, NY et en ligne du 17 au 20 octobre 2022. La soumission est prévue pour le lundi 17 octobre à 16h30 HNE (UTC-04:00).
approche généralisable
Bien que de nombreux réseaux de neurones aient été développés pour accomplir la tâche lourde de données de reconstruction d’hologrammes recherche biologique Et le Applications biomédicalesLa plupart sont conçus pour être très spécifiques. Cela signifie qu’il peut ne pas fonctionner correctement s’il est utilisé avec des échantillons autres que ceux initialement utilisés pour la formation réseau.
Pour résoudre ce problème, Chen et ses collègues ont développé un réseau neuronal complet appelé Fourier Imager Network (FIN). Ce type de réseau de neurones est formé à l’aide d’un modèle unique, en contournant certaines étapes généralement utilisées par d’autres méthodes d’apprentissage en profondeur. Les réseaux de neurones de bout en bout sont également plus rapides et plus généralisables à une variété d’échantillons.
Des résultats plus rapides et plus précis
Le cadre FIN prend une série d’hologrammes de densité brute uniquement pris à différentes distances de l’échantillon au capteur à l’aide d’un microscope 3D sans lentille intégré et crée des images reconstruites des échantillons. Pour tester la nouvelle approche, les chercheurs ont entraîné le réseau à l’aide de sections de tissu pulmonaire. Ils ont ensuite utilisé FIN pour reconstruire des images 3D de tissus de glandes salivaires humaines et d’échantillons de frottis Pap que le réseau n’avait pas vus pendant la formation.
FIN a bien fonctionné sur ces nouveaux types d’échantillons et a fourni des images qui ont été reconstruites avec plus de précision qu’un algorithme itératif et un modèle moderne d’apprentissage en profondeur. Il a également montré une vitesse améliorée de près de 50 fois par rapport au modèle d’apprentissage en profondeur. Les chercheurs affirment que ces résultats démontrent la forte généralisation extrinsèque de FIN tout en démontrant l’énorme potentiel de la construction profonde d’un généralisable à grande échelle. les réseaux de neurones Pour diverses tâches de microscopie et d’imagerie computationnelle.
Chen a ajouté : « Notre prochaine étape consiste à étudier l’AF tout en conservant les avantages de notre approche, tels qu’une superbe qualité d’image, une généralisation sans précédent de nouveaux types d’échantillons et une vitesse de calcul améliorée, rendant l’imagerie holographique possible avec un matériel à faibles ressources.
la citation: Neural Network Speeds for Holographic Image Reconstruction of Biological Samples (2022, 22 septembre) Récupéré le 22 septembre 2022 sur https://phys.org/news/2022-09-neural-network-holographic-image-reconstruction.html
Ce document est soumis au droit d’auteur. Nonobstant toute utilisation équitable à des fins d’étude ou de recherche privée, aucune partie ne peut être reproduite sans autorisation écrite. Le contenu est fourni à titre informatif uniquement.





