Le rôle des bactéries dans la croissance du cancer
Le microbiote associé aux tumeurs est un élément important du microenvironnement tumoral (TME) dans 33 cancers humains. Cependant, peu de preuves sont disponibles concernant la distribution spatiale et la localisation de ces microbes dans les cellules tumorales.
Combler cette lacune de la recherche est récent la nature Une étude périodique a évalué les interactions spatiales, cellulaires et moléculaires du microbiote dans le carcinome épidermoïde oral (CCSO) et le carcinome colorectal (CRC). Dans cette étude, les scientifiques ont cartographié les interactions cellulaires-bactériennes, spatiales et moléculaires au sein du TME en utilisant le séquençage d’ARN unicellulaire (scRNA-seq) et Sur site techniques de profilage spatial.

Contexte
Les tumeurs des patients cancéreux sont généralement constituées de cellules malignes entourées d’un réseau complexe de cellules non malignes. Ces cellules peuvent présenter des effets pro- ou anti-tumoraux selon leur abondance et leur type. Tous les deux dans le laboratoire Et le in vivo Des expériences ont indiqué la présence de bactéries dans le microbiote associé aux tumeurs, qui jouent un rôle important dans le développement du cancer, la surveillance immunitaire, les métastases et la chimiorésistance. L’analyse moléculaire et les données de bioimagerie ont également démontré la présence de microbiote intratumoral dans les principaux types de cancer.
Il existe un manque de preuves concernant l’identité spécifique des cellules hôtes par lesquelles les microbes associés aux tumeurs interagissent avec les cellules tumorales des patients cancéreux. De plus, peu de preuves concernant l’identification de cellules spécifiques hébergeant des organismes ont été documentées. L’influence des interactions cellulaires subtiles entre l’hôte et les microbes et la distribution spatiale des microbes au sein de la tumeur sur leurs capacités fonctionnelles au sein du TME n’est pas claire.
À propos de l’étude
Le séquençage du gène de l’ARNr 16S des tissus tumoraux des patients atteints de CCR a indiqué la présence de diverses bactéries, y compris broche. L’abondance de ces bactéries différait chez les patients atteints de CCR. Le dendrogramme et l’analyse des composants principaux avec la cohorte de diversité bêta ont indiqué que la majorité des patients atteints de cancer avaient des compositions de microbiome relativement stables. Cependant, la plupart des patients ont montré divers degrés d’hétérogénéité dans la composition du microbiome au sein de la tumeur.
Le RNAscope-fluorescence Sur site L’imagerie d’hybridation (RNAscope-FISH) a confirmé la distribution spatiale hétérogène des communautés bactériennes dans le TME. Les données basées sur RNAscope-FISH ont montré qu’il y a broche Noyauqui a été validé par l’analyse du microbiome et la technologie quantitative de réaction en chaîne par polymérase.
Le transcriptome spatial 10x Visium a également été utilisé pour détecter et analyser la distribution spatiale des microbes dans la tumeur des échantillons de CRC et d’OSCC. Cette approche a identifié 28 % des taches capturées dans les tumeurs OSCC et 46 % des tumeurs CRC.
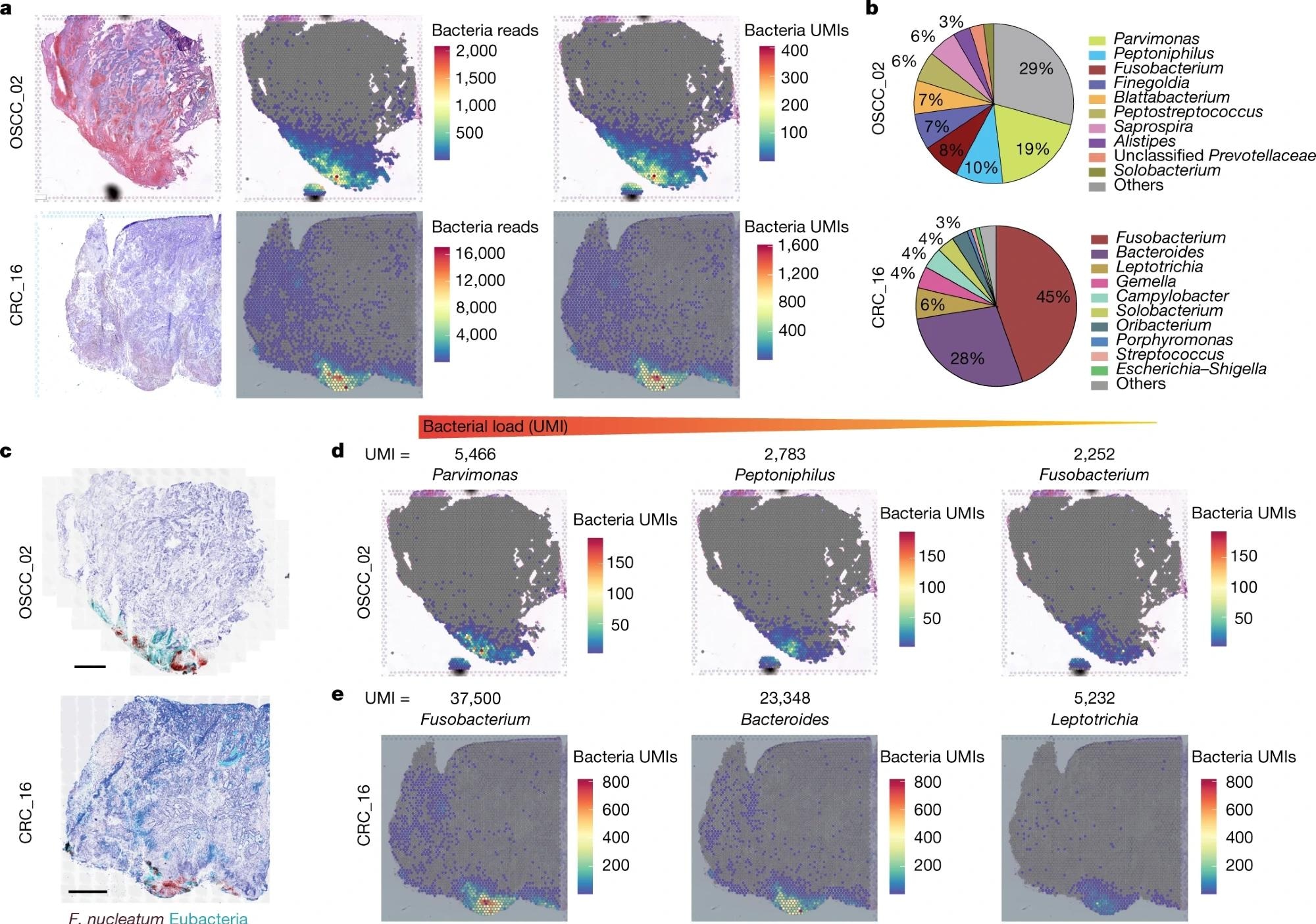
dans une tumeur OSCC, ParphémonasEt le peptonephilusEt le broche Des souches dominantes ont été trouvées, tandis que broche Et le Bacteroides Les races tumorales étaient dominantes dans le CCR.
La technologie spatio-temporelle 10x Visium a permis la détection directe, la quantification et la cartographie spatiale des bactéries viables dans les tissus tumoraux sains des patients atteints de cancer. Il a également indiqué la complexité des interactions des microbes dans la tumeur à travers le tissu tumoral.
La plateforme de profilage spatial numérique (DSP) GeoMx a permis de déterminer le profil d’expression de 77 protéines associées au développement du cancer et à l’immunité anti-tumorale. Cette technique, associée aux approches RNAscope et immunohistochimie (IHC), a indiqué que les communautés bactériennes habitent des sutures très fines qui sont immunosuppressives et peu vasculaires. De plus, les souches bactériennes ont tendance à habiter les cellules malignes avec de faibles niveaux de Ki-67.
La technique INVADEseq (Invasion- and Adhesion-Directed Expression Sequencing) a été développée pour évaluer l’interaction bactérienne hôte-cellule-cellule au sein du TME et l’effet sur la transcription de la cellule hôte. Cette technique est associée à l’insertion d’une amorce pour cibler les régions conservées de l’ARNr 16S bactérien. Par la suite, des bibliothèques (ADNc) avec des transcrits bactériens peuvent être produites à partir de cellules humaines associées à des bactéries. Un aspect critique de cette méthode est que l’insertion d’amorce n’affecte pas le profil d’expression génique des cellules CRC humaines.
La technique INVADEseq a été validée en utilisant la lignée cellulaire de CRC humain HCT116 qui a été infectée par deux espèces bactériennes, à savoir : F. NoyauEt le Porphyromonas gingivalisEt le Prévotelle intermédiaire. permis la cartographie des populations bactériennes dans des cellules humaines individuelles. Fait important, INVADEseq a confirmé un rôle F. Noyau Et le s. gencives affectant l’hétérogénéité des cellules cancéreuses. Ces souches bactériennes modifient des programmes de transcription distincts qui facilitent le regroupement cellulaire spécifique. L’altération des voies transcriptionnelles est également associée à des manifestations d’inflammation, de dormance cellulaire, de métastases et de réparation de l’ADN.
conclusions
La présente étude a révélé que les cellules tumorales infectées par des bactéries affectent leur environnement comme une seule cellule, qui recrute ensuite des cellules myéloïdes dans la zone bactérienne. Notamment, le microbiome présent dans la tumeur n’était pas un phénomène aléatoire. Au lieu de cela, la présence de bactéries dans une tumeur a été considérée comme un processus hautement régulé dans des microniches avec des fonctions cellulaires immunitaires et épithéliales affectant le développement du cancer.
Bien que l’étude actuelle se soit concentrée sur deux types de cancer, les outils et les techniques peuvent être utilisés pour étudier tous les principaux types de cancer et ceux avec des microbes dans la tumeur.





